北京大学数学科学学院/统计科学中心席瑞斌课题组近期在著名期刊《Experimental&Molecular Medicine》(EMM,影响因子=12.15)上发表了“The shaping of cancer genomes with the regional impact of mutation processes”的研究论文。该研究突破了现有突变指纹(mutation signature)分析均在全基因组水平上进行的局限,发展了一种基于贝叶斯信息准则(BIC)的对基因组变异进行分区、聚类的统计方法;通过对每个聚类区域进行局部突变指纹分析,计算了每个点突变由特定突变指纹产生的后验概率,进而可对每个点突变进行突变过程的溯源,促进对癌症发展的分子机制、癌症基因组的进化历史的理解。

突变指纹是由特定突变过程引起的突变类型的特征性组合,例如在与紫外线照射相关的突变指纹中,会出现很多与T>C 转换相关的突变类型。通过对基因组突变数据进行非负矩阵分解,人们已经发现了与紫外线照射、吸烟、酒精代谢、APOBEC 酶活动、基因组错配修复缺陷等因素相关的30 多种突变指纹。目前的突变指纹分析均在全基因组的水平上进行,然而,DNA 损伤、修复和复制等过程存在区域不一致性,导致突变指纹在基因组上的分布也有区域异质性。由于区域异质性的存在,基于基因组全局的突变指纹分析,会导致局部发生的突变过程对应的突变指纹的错误判断。因此发展相应的统计方法将基因组中突变分布相似的区域进行聚类,并进行局部突变指纹分析是非常有必要的。
针对这一挑战,文章发展了一种基于BIC的对基因组变异进行分区、聚类的统计方法,
此方法将基因组中突变进行空间上的分区,确定基因组上突变分布发生显著改变的位置,再对这些分区进行聚类,将突变类型的分布相似的区域聚类到一起,最后基于得到的每个聚类区域进行局部突变指纹分析。文章对约2000个来自ICGC数据库的癌症基因组进行了大规模分析,对约50,000,000个点突变进行了突变指纹的溯源。
经过后续生物信息学分析,文章得到了很多新发现——与 DNA复制/转录链不对称相关的突变指纹显示出明显的遗传/表观遗传特征;虽然与已知的癌症驱动基因相关的突变指纹是多样化的,但该研究观察到几乎一半的在氨基酸水平上的突变热点具有单一的突变指纹;在TP53上有多个突变指纹聚集,这表明该基因是各种突变过程的通用靶标;发生在kataegis(癌症基因组中的局部超突变现象)区域中的突变可能由多个突变过程引起的。文章进一步从进化的角度对突变进行了解剖,在关于突变特征的进化轨迹的研究中,揭示了多个突变过程在对癌症基因组进行重塑时的独特进化时间尺度。
北京大学席瑞斌和The Catholic University of Korea的Tae-Min Kim为本文的共同通讯作者,北京大学统计科学中心15级博士生王涵和The Catholic University of Korea的Soo-Youn Lee博士为本文的共同第一作者。
论文链接:
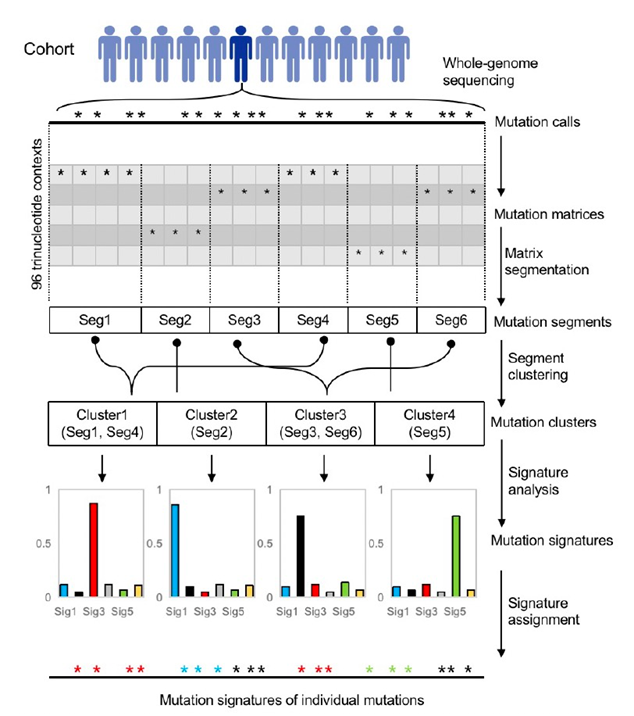
图一:方法流程图
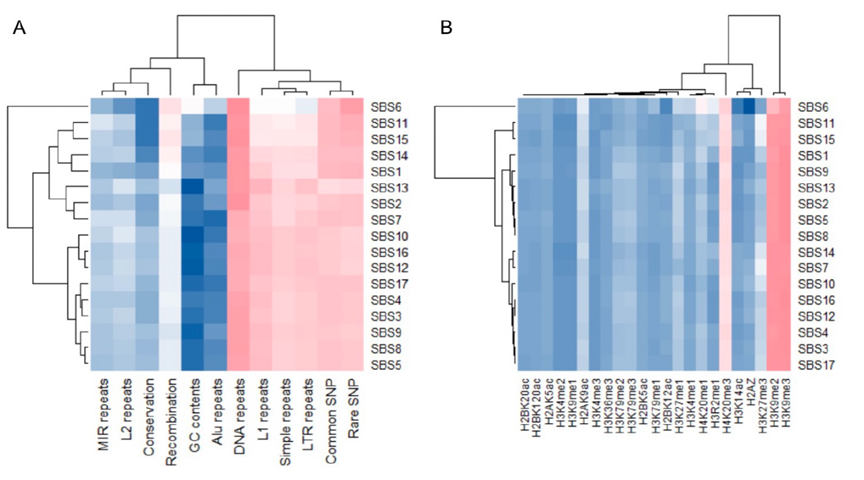
图二:突变指纹的基因组学、表观遗传学联系
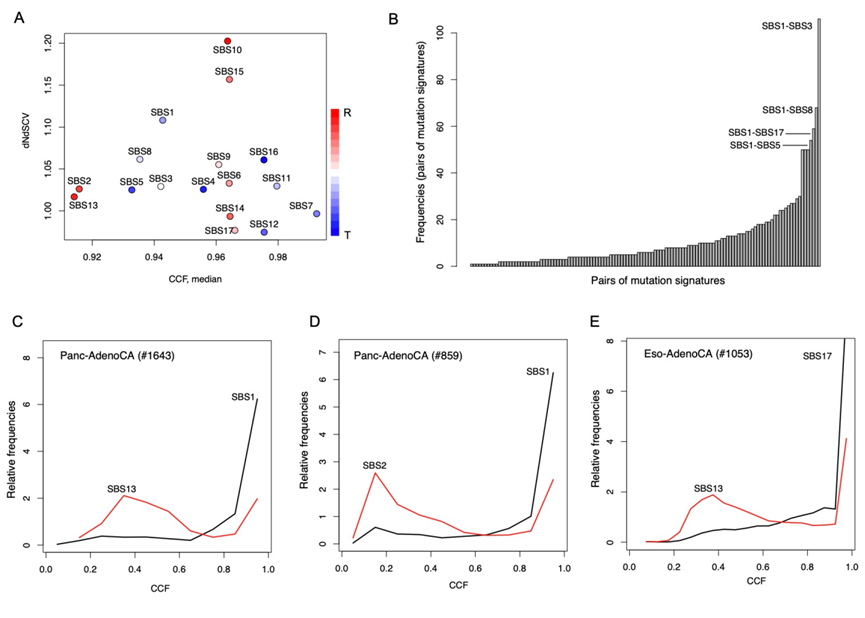
图三:突变指纹的进化分析


